酶标仪利用”无创”技术检测活细胞荧光蛋白
简介
在过去的五年中,荧光蛋白在监测体内生物学研究中,起到越来越重要的作用。源于维多利亚多管发光水母中的绿色荧光蛋白(GFP)是最早被我们应用的荧光蛋白,但是随着时间的推移,现在我们可以使用的荧光蛋白种类也越加丰富,包括加强型的变异GFP蛋白、从其他种类水母中发现的荧光蛋白和珊瑚礁蛋白。它们都可以在众多的细胞和组织中被克隆复制,从细菌到酵母,从植物到哺乳动物。这些荧光蛋白稳定、细胞毒性小、而且不需要额外的辅助也能在体内产生可见的荧光。因此,它们可以被用作分子靶标或者独立的报告因子去视化、追踪和定量多种不同的细胞进程,包括细胞合成与代谢,蛋白转运,基因诱导和细胞谱系。不同的荧光蛋白具有不同的颜色,因此也可被用于多重检测。这些荧光蛋白都能被荧光显微镜和流式细胞仪检测到。但是相应的,如果我们不做细胞分选或监测细胞内迁移,微孔板荧光读板仪将是一个更好、更方便和更高通量的检测系统。下面,我们将会向大家展示Molecular Devices酶标仪如何利用”无创”技术检测活细胞荧光蛋白。
我们将从BD Biosciences Clontech公司获得的三株HEK-293细胞系进行不同荧光蛋白的稳定转染。这次研究的目的:1)优化三株细胞系的波长设置,2)通过稀释细胞系来确定检测下限LLD,3)证明在一种细胞系中识别另一种细胞系的可行性。
材料:
HEK-293细胞系稳定表达三种荧光蛋白:
AcGFP---多管发光水母中产生的另一种GFP(但不同于维多利亚水母)
ZsGreen---与GFP相似,但更亮(来自珊瑚礁)
DsRed---一种来自珊瑚礁的红色荧光蛋白非转染的HEK-293细胞系,作为对照
方法:
细胞准备与分析
细胞都培养在大烧瓶中,应用含10% FBS+ 1% Pen/Strep/L-glutamine +500 μg/mL G 418的DMEM的培养基。没有转染的HEK-297细胞作为对照。在实验前一夜,对细胞进行胰酶消化,然后进行密度梯度稀释,最终密度范围为从500,000到100个细胞每毫升。然后把它们过夜接种到96孔(100ul)和384孔(25ul)板中。因此,接种的细胞密度就是50,000到10个细胞每孔(96孔板)和12,500到2.5个细胞每孔(384孔板)。每个稀释都做12个重复。第二天分别用SpectraMax M5和Gemini EM两台仪器对酶标板进行底读和顶读检测。
波长优化
SpectraMax M5和Gemini EM都是基于单色器的扫描式荧光读板仪。针对每一个特殊荧光染料,其激发波长和发射波长都可以在背景之上进行信号优化,这点也是滤光片式酶标仪所不具备的。总的来说,优化策略就是:用低于理论最大激发波长20-25nm的激发光激发,然后初扫发射波长;其次用高于理论最大发射波长20-25nm的发射光扫激发波长。扫描后将给出实际的激发与发射波长。最后可以结合信号/背景分析再做其他额外的扫描来确定最终的结果。(根据斯托克顿位移理论,激发波长应该更低、发射波长应该更高,并且需要一个发射光阻隔滤片来隔离掉非目的波长的光信号)。而且可能还会因为要选择最佳阻隔滤片而再进行两次扫描。
结果:
波长优化
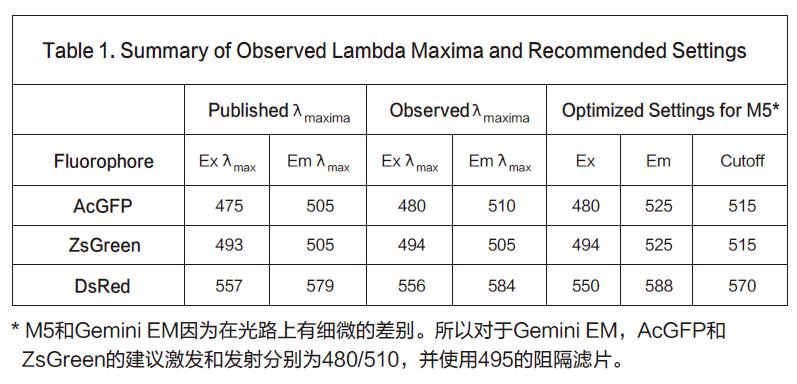
用Gemini EM对DsRed进行波长扫描,以此举例如何进行波长优化。为了检测最大激发波长, 我们首先固定发射波长为600nm,然后对发射波长进行扫描。扫描结果显示最大激发波长为556nm。(图1)同样为了检测最大发射波长,我们固定激发波长为535nm,然后扫描发射波长,从而得到584nm的结果。(图2)激发与发射值都与被发表的波长结果557/579相近。
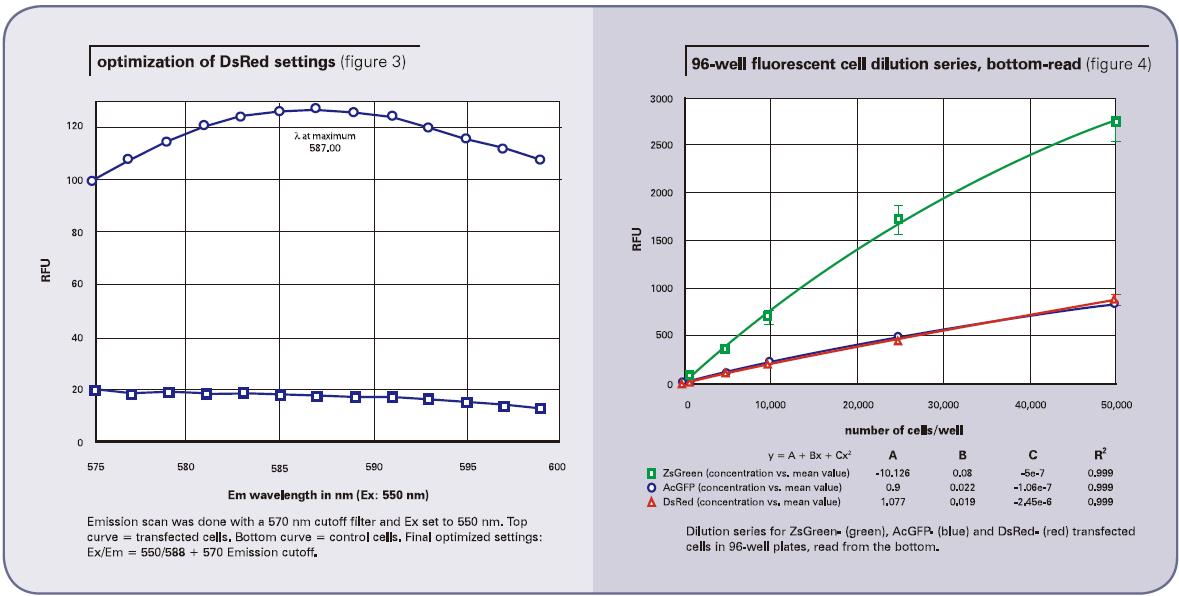
下一步就是要结合信号/背景优化结果确定最佳激发和发射波长。因为初步检测结果的斯托克顿位移偏小(22nm),显然是要通过降低激发波长和增大发射波长来扩大两者之间的差异,其次还需要找到合适的发射光阻隔滤片优化最佳灵敏度。最终,我们使用5 5 0nm的激发波长来激发, 同时使用570nm的发射光阻隔滤片隔离掉不想要的高于570nm以上的激发光。通过575到600nm的发射波长扫描DsRed转染的细胞,结果显示在587-588nm处会有峰值出现(图3,上面的曲线)。背景(没有转染的细胞)区域中,点就相对比较平缓(图3,下面的曲线)。基于本次扫描,我们最终优化的设置为550nm激发、588nm发射、575nm作为发射光阻隔滤片。
观察到的最大值与我们建议用于定性分析的设置都总结在列表1中。
细胞稀释的结果
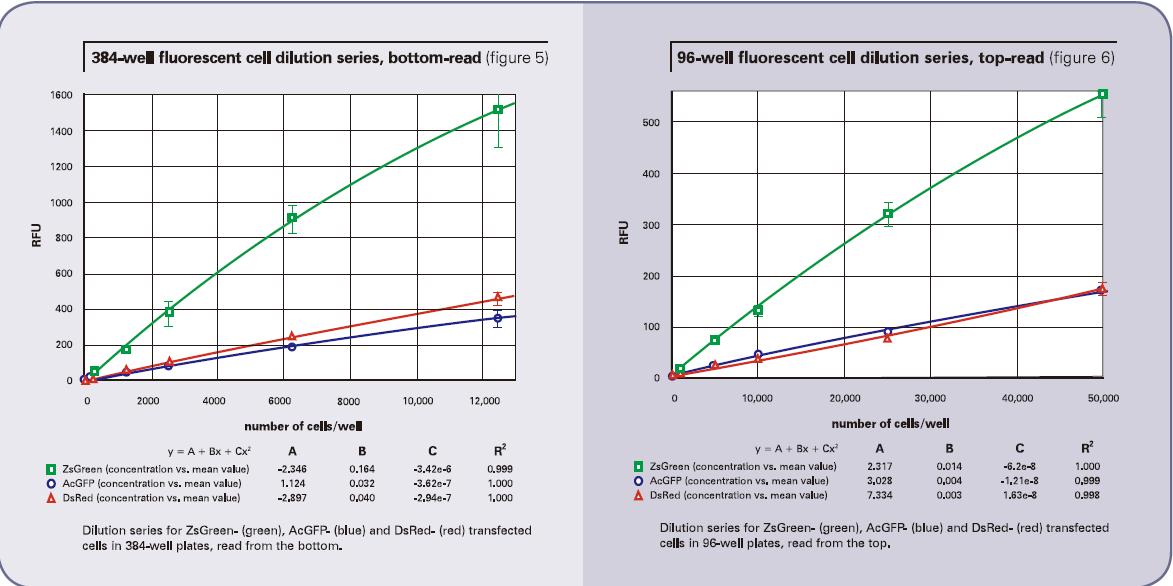
采用底读得到的细胞稀释结果详见图4(96孔板)和图5(384孔板)。顶读结果详见图6(96孔板)和图7(384孔板)。ZsGreen细胞系(上面的曲线)的亮度是其它两种细胞系的将近3.5倍。尽管DsRed细胞系的结果暗于ZsGreen,但两种细胞系的检测灵敏度却相近。(列表2的下图),因为背景值低于DsRed的波长。
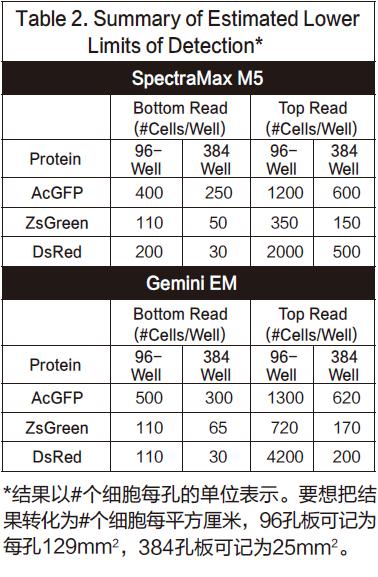
总的来说,图中的点稍微有些非线性,最上方会有一点下降。我们推断是因为细胞在高浓度时,会有贴壁生长的趋势,所以会造成非线性的结果。列表2给出了所有细胞系确认得到的检测下限L L D 。其中L L D 的计算方法是:3SDBLANK/Slope,SDBLANK是背景孔(没有细胞的培养基)的标准差,Slope是曲线底部的斜率。(对于96孔板和384孔板,分别使用了10,000个细胞每孔和2500个细胞每孔的数据)。非转染细胞的RFU信号和标准差结果都与只含有培养基的结果相似。
在本次实验中,ZsGreen和DsRed细胞系在底读模式都有着相似的检测限,而且两者都比AcGFP细胞系的检测下限低3到4倍。本次实验一共重复了三次,但是DsRed实验结果并不是每次都能表现的足够好。在一次实验中,它的检测下限近似于AcGFP,但是在另一次实验中,它的检测下限又会很高。我们将这些区别归结于不同细胞系的通道数目不同。(伴随着一个成功的通道,细胞就会变得越暗)DsRed细胞系在实验开始阶段比其它细胞系长得快,所以在第一次实验中,它比其它细胞系多2-4倍的通道,结果就显然不够理想。
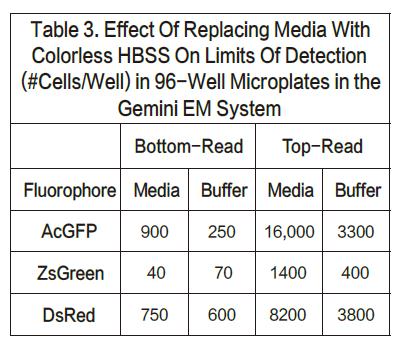
对于AcGFP和ZsGreen细胞系,当采用顶读时,检测下限升高将近三倍。另一方面,对于DsRed,当采用顶读时,检测下限会升高将近10-20倍。我们认为造成这种灵敏度下降的原因在于DMEM培养基有红色荧光信号,会对结果有内部干扰。事实也证明,将培养基换成无色的HBSS,相应的DsRed的顶读结果也会变好。列表3展示了将有色培养基换成无色培养基的结果。底读并没有太多改变(尽管AcGFP的检测下限看起来改善了),证明这种操作方法并没有导致细胞的损失。然而顶读结果却让灵敏度改善了将近三倍。这些结果也从某一方面支持了一种理论,那就是有色培养基会一定程度上影响顶读的灵敏度。
细胞混合结果
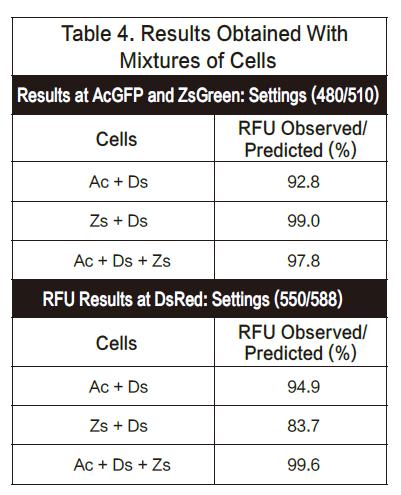
在本次实验中,我们在一个96孔板里混合了多种细胞,保证每个孔大约50,000个细胞。因此,如果是1:1混合,那么每种细胞会有25,000个。如果是1:1:1混合,那么每种细胞将有1 6 , 7 0 0 个。实验板分别用480/510nm(AsGFP和ZsGreen的优化结果)和550/588nm(DsRed设置)的激发和发射来检测。观察到的RFU值与预测值接近。结果总结如下。结果显示,AsGF P和ZsGreen可以和DsRed一起检测,反之亦然。(见图表4)
结论
Molecular Devices公司的荧光读板仪可以检测完整贴壁细胞中的荧光蛋白。波长扫描可调的功能可以优化每一种独特荧光染料的波长结果。底读模式提供最佳检测灵敏度,顶读模式也可以提供可利用的数据,尤其是对含有ZsGreen和DsRed的细胞系。顶读模式检测DsRed细胞系时,可以通过替换无色培养基来达到优化实验结果的目的。
【参与有礼】美谷分子微孔板使用调研
(2024-09-11T00:00 浏览数:30011)
【订购有礼】美谷分子电商产品上线啦!
(2024-09-11T00:00 浏览数:29972)
【webinar预告】肝脏类器官研究进展及应用分享
(2024-09-10T00:00 浏览数:30889)
【Webinar 预告】中药活性成分的生物合成探索之路
(2024-06-21T00:00 浏览数:47840)
【多重好礼】618电商狂欢盛典 限时福利大放送!
(2024-06-18T00:00 浏览数:35755)
【会议预告】类器官 3D 培养与高内涵成像应用论坛—西安站
(2024-06-07T00:00 浏览数:37183)
【注册有礼】入驻丹家智选商城 美谷分子邀您体验云采购
(2024-05-27T00:00 浏览数:35626)
【会议预告】高内涵成像与类器官3D体外模式智能化培养用户会议广州站
(2024-05-16T00:00 浏览数:36675)
美谷分子入驻丹家智选商城 开启全新购物体验
(2024-04-29T00:00 浏览数:38382)
【Webinar预告】合成生物学前沿应用:酶工程
(2024-04-12T00:00 浏览数:35776)